Los investigadores han descubierto un nuevo grupo de toxinas bacterianas que pueden matar bacterias y hongos dañinos, abriendo la puerta a posibles nuevos tratamientos para las infecciones. Estas toxinas, que se encuentran en más de 100.000 genomas microbianos, pueden destruir las células de bacterias y hongos sin dañar a otros organismos.
Un nuevo estudio, publicado en Nature Microbiology, dirigido por los investigadores de la Universidad Hebrea, el Dr. Asaf Levy del Instituto de Ciencias Ambientales, la Dra. Neta Schlezinger de la Escuela de Medicina Veterinaria, y el Dr. Netanel Tzarum del Instituto de Ciencias de la Vida, en colaboración con los investigadores del Instituto Weizmann de Ciencias, los Profs. Jacob Klein y Meital Oren-Suissa, junto con el profesor Herbert Schmidt de la Universidad de Hohenheim, han revelado un nuevo arsenal de toxinas bacterianas con el potencial de combatir enfermedades infecciosas humanas y vegetales. Estas toxinas, que están codificadas en los genomas de ciertas bacterias, exhiben potentes propiedades antibacterianas y antifúngicas, lo que ofrece nuevas y emocionantes posibilidades para aplicaciones clínicas y biotecnológicas.
La competencia microbiana es un fenómeno natural, y las bacterias han desarrollado métodos sofisticados, incluidas toxinas, para eliminar a los competidores. Los ejemplos más famosos de compuestos naturales utilizados en competición en la naturaleza son los antibióticos producidos por bacterias y hongos.
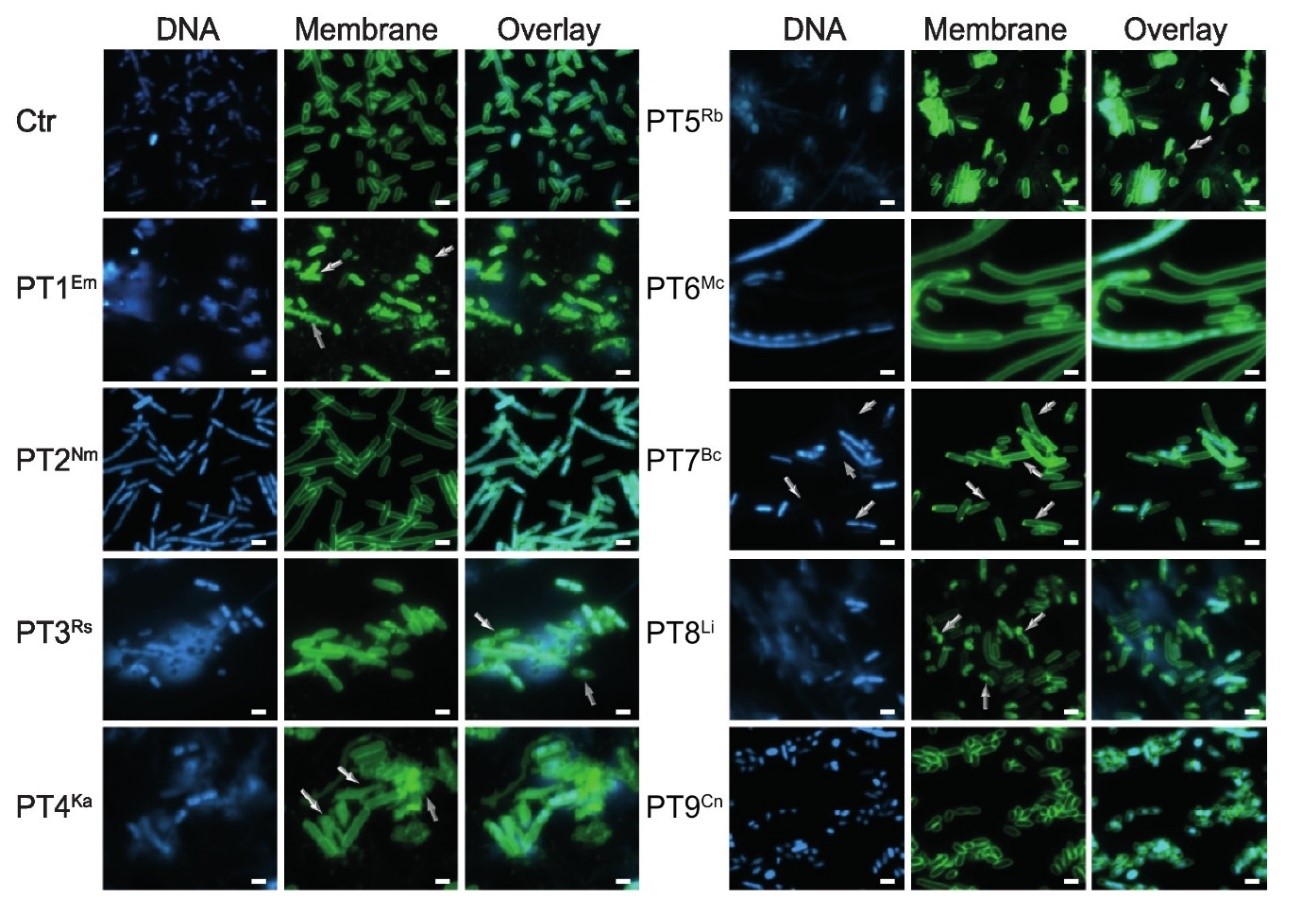
En este estudio, el equipo del Dr. Levy desarrolló un enfoque computacional innovador para identificar dominios de proteínas de toxinas no descubiertos anteriormente, que tienen entre 100 y 150 aminoácidos de largo, dentro de más de 105.000 genomas microbianos. Estas toxinas proteicas, denominadas toxinas polimórficas, desempeñan un papel crucial en la guerra microbiana, ya que apuntan y matan a los microorganismos que compiten en diferentes ecosistemas.
El equipo de investigación de los estudiantes y becarios postdoctorales: Nimrod Nachmias, Noam Dotan y la Dra. Marina Campos Rocha, y las investigadoras Dra. Yaara Oppenheimer-Shaanan y Rina Fraenkel, validaron con éxito nueve toxinas recién descubiertas, cada una de las cuales representa una gran familia conservada evolutivamente, demostrando su capacidad para causar la muerte celular tanto en Escherichia coli como en Saccharomyces cerevisiae cuando se expresa en estos organismos modelo.
Cabe destacar que también se identificaron cinco genes de antitoxina, también conocidos como genes de inmunidad, que protegen a las bacterias produciendo las toxinas de la autodestrucción.
Es interesante notar que las toxinas exhiben una poderosa actividad antifúngica contra una variedad de hongos patógenos, mientras que dejan sin afectar a ciertas especies de invertebrados y macrófagos. Los resultados experimentales del estudio sugieren que estas toxinas actúan principalmente como enzimas eficientes que se dirigen a procesos celulares esenciales, como la membrana celular, el ADN o la división celular. El análisis estructural de dos complejos proteicos de inmunidad a toxinas confirmó aún más que algunas de estas toxinas poseen actividad de DNasa, que puede degradar el ADN en las células diana. Curiosamente, la estructura muestra que la toxina está cargada positivamente en su sitio de unión al ADN, para unirse al ADN cargado negativamente, mientras que la proteína de la antitoxina está cargada negativamente para evitar que la toxina se una al ADN objetivo.
“Nuestros hallazgos amplían nuestra comprensión de cómo las bacterias usan toxinas en competencia con otros microbios y proporcionan vías emocionantes para futuras investigaciones sobre agentes antimicrobianos críticamente necesarios contra patógenos bacterianos y fúngicos humanos y vegetales. El potencial de estas toxinas para servir como base para nuevos tratamientos clínicos o innovaciones biotecnológicas es particularmente emocionante”.
Dr. Asaf Levy, Universidad Hebrea de Jerusalem
Esta investigación no solo mejora el conocimiento de las toxinas microbianas, sino que también arroja luz sobre su posible uso terapéutico. El descubrimiento del equipo podría allanar el camino para nuevas estrategias antimicrobianas, especialmente a medida que el mundo se enfrenta al aumento de patógenos resistentes a los antibióticos.
El estudio tiene amplias implicancias tanto para la comprensión de las interacciones microbianas en diferentes entornos como para el desarrollo de antimicrobianos de próxima generación. Al revelar los mecanismos a través de los cuales operan estas toxinas, la investigación ofrece esperanzas para nuevos tratamientos en la lucha continua contra las infecciones bacterianas y fúngicas.
El artículo de investigación titulado “Systematic Discovery of Antibacterial and Antifungal Bacterial Toxins”, ya está disponible en Nature Microbiology.
Investigadores:
Nimrod Nachmias1, Noam Dotan1, Marina Campos Rocha2, Rina Fraenkel3, Katharina Detert4, Monika Kluzek5, Maor Shalom1, Shani Cheskis1, Sonu Peedikayil-Kurien6,7, Gilad Meitav1, Arbel Rivitz1, Naama Shamash-Halevy1, Inbar Cahana3, Noam Deouell3, Jacob Klein5, Meital Oren-Suissa6,7, Herbert Schmidt4, Neta Schlezinger2, Netanel Tzarum3, Yaara Oppenheimer-Shaanan1, and Asaf Levy1.
Instituciones:
1) Departamento de Patología Vegetal y Microbiología, Instituto de Ciencias Ambientales, Facultad Robert H. Smith de Agricultura, Alimentación y Medio Ambiente, Universidad Hebrea de Jerusalem.
2) Escuela Koret de Medicina Veterinaria, Facultad Robert H. Smith de Agricultura, Alimentación y Medio Ambiente, Universidad Hebrea de Jerusalem.
3) Departamento de Química Biológica del Instituto Alexander Silberman de Ciencias de la Vida, Facultad de Ciencias y Matemáticas, Universidad Hebrea de Jerusalem.
4) Instituto de Ciencia y Biotecnología de los Alimentos, Departamento de Microbiología de los Alimentos, Universidad de Hohenheim, Stuttgart, Alemania.
5) Departamento de Materiales e Interfaces, Instituto Weizmann de Ciencias.
6) Departamento de Ciencias del Cerebro, Instituto Weizmann de Ciencias.
7) Departamento de Neurociencia Molecular, Instituto Weizmann de Ciencias.


